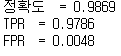사용한 DataFrame

1.molecular fringerfrint
2. Physchem properties
-MolWight, LogP, MP, BP, LogVP, LogWS, LogHL, LogKOA, LogD55, LogD74, LogOH, RT
3. Environmental fate
- LogBCF, ReadyBiodeg,LogKM, LogKOC
6개 assay에 대해서 active(1) inactive(0) 예측 모델을 만든다.
모든 assay에 대하여 아래와 같은 모델과 검증 방법을 사용한다.
fingerfrint만 사용했을때 vs featrue를 추가했을때



confustion matrix를 읽는 방법은
실제정답
| 0(inactive) | 1(active) | |
| 0(inactive) | conf_mat[0][0] : TP | conf_mat[1][0] : FP |
| 1(active) | conf_mat[0][1] : FN | conf_mat[1][1] : TN |
acc = TP+TN / TP + FN + FP + TN
TPR(sensitivity,recall) = 맞춘양성(0)/실제양성(0) = TP /TP + FN
FPR = 틀린음성/실제음성 = FP / FP + TN
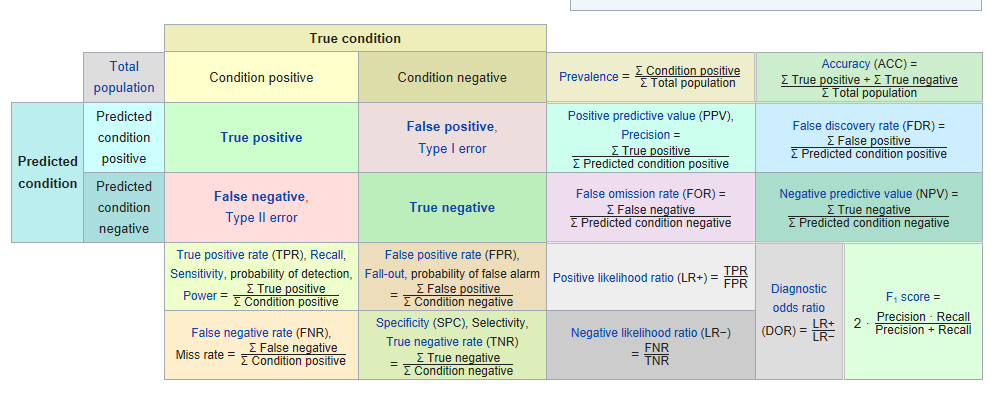
TOX21_AhR_LUC_Agonist
fingerfrint만 사용했을때


15개의 feature들을 전부 추가했을때


오히려 성능이 떨어졌다. --> tpr에 꽤 영향을 미쳤다
평가 방식 자체에 문제가 있다.
duplicate chemical들을 살펴보자.
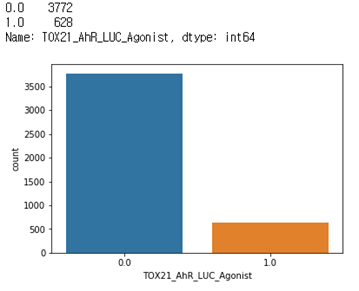
TOX21_ERa_BLA_Agonist_ratio
fingerfrint만 사용했을때


feature를 추가했을때

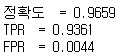
FP는 증가하고 FN은 감소했다.
TOX21_ERa_LUC_BG1_Agonist
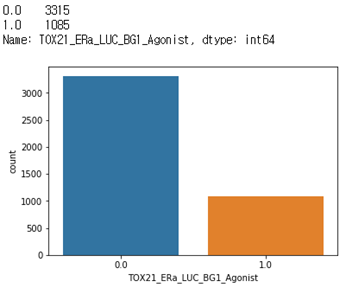
fingerfrint만 사용했을때

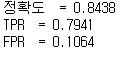
feature를 추가했을때
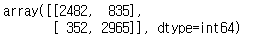

input : 2048dim
random forest 결과는?
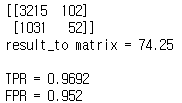
input : 10dim(pca로 축소)

트리를 76->200개로 늘려도

input : 2063dim(feature 추가)

트리를 76 -> 500개로 늘려도

input : 2048dim(fingerfrint only)
smote를 적용시킨 후에 분류성능이 굉장히 올랐다.(그러나 실제성능이 아니다)

input : 2063dim(feature added)

input : 512dim(fingerfrint only), allsmote
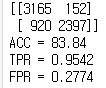
input 527dim(feature added), all smote

svm
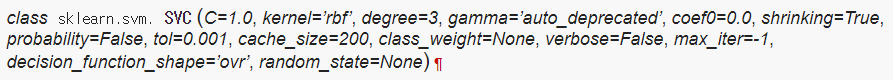


SVM : input (15 features) all smote
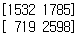

RF : input(15features) all smote


RF : input(15features) no smote

input : 527 ,RF, undersampling

TOX21_PPARg_BLA_Agonist_ratio
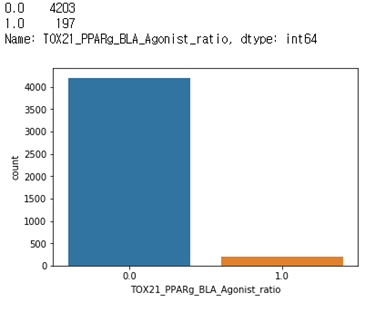
fingerfrint만 사용했을때
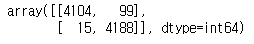

feature를 추가했을때